EGFR/EGFRvIII-Amplifikation bei diffusen IDH-Wildtyp Astrozytomen
Eine Vervielfachung (Amplifikation) des Epidermal growth factors EGFR und dessen verkürzter, aber dauer-aktivierter Variante EGFRvIII tritt häufig in Glioblastomen (GBMs) auf. Deren Vorkommen spielt eine kritische Rolle in der Pathogenese des Tumors und dessen Progression und geht mit einer erhöhten EGFR-Proteinmenge einher (1,2). Bei primären GBMs tritt in knapp 60% der Fälle eine EGFR-Amplifikation auf, bei sekundären lediglich in 8% der Fälle (2). Die EGFRvIII-Variante wird dabei im späten Krankheitsverlauf häufiger beobachtet und entsteht durch Verluste von Exon 2 bis Exon 7 des Wildtyp-EGFRs, vermutlich nach der eigentlichen Vervielfältigung des Gens (3). Gemäß der aktualisierten WHO-Richtlinien von 2021 werden diffuse Astrozytome ohne IDH-Mutation (IDH-wild type diffuse astrocytic) bei Vorliegen einer EGFR-Amplifikation, einer TERT-Promotor-Mutation, und/oder eines Zugewinnes von Chromosom 7 bei gleichzeitigem Verlust von Chromosom 10 (+7/-10) als Astrozytome Grad 4 (= GBM) gradiert, auch wenn sie histologisch nur als WHO Grad 3 oder niedriger erscheinen, da sie einen aggressiveren Verlauf aufweisen als ohne diese genetischen Veränderungen (4).
Dementsprechend ist eine Bestimmung der Kopienzahl des EGFR-Gens und ggfls. das Vorliegen der Variante zur exakten Diagnose vor allem niedriggradiger Astrozytome notwendig. Dafür nutzen wir eine einfache und robuste quantitative real-time-PCR-Methode. Sie nutzt den DNA-interkalierenden Farbstoff SYBR Green, um Veränderungen der EGFR-Kopienzahl, relativ zu einem Referenzgenom (EDTA-Vollblut des/der Patient*in) und normalisiert auf zwei Referenz-Genloki zu quantifizieren (5,6). Die Cq-Werte der EGFR-Gen-Abschnitte von Exon 3, Exon 17, und Exon 22 (Chromosom 7), PGM5 (auf Chromosom 9) sowie von β-Actin und B2M werden ermittelt und repräsentieren dabei die Gendosis in der Tumor- und der Blutprobe. Die letzteren beiden (β-Actin und B2M) sind probeninterne Referenzen (ΔCq), um Qualitäts- und Mengenunterschiede der DNA zu eliminieren. Für etwaige Vervielfältigung, hervorgerufen durch somatische Veränderungen im Tumor (die nicht im Blut vorhanden sind), wird die Differenz der Cq-Werte zwischen Tumor- und Blutprobe bestimmt (ΔΔCq). Ist das relative Verhältnis des EGFR-Gens in Tumor/Blut gleich 1, liegt keine Vervielfältigung vor. Bei einem Verhältnis von 1,5 liegt eine mögliche Trisomie von Chromosom 7 vor. Bei Werten über 2 liegt eine lokale Vervielfältigung des EGFR-Genabschnittes vor. PGM5 wird mitbestimmt, da Chromosom 9 an der Position des PGM5-Gens (chr9 q21.11) eher selten in glialen Tumoren chromosomale Anomalien aufweist (7).
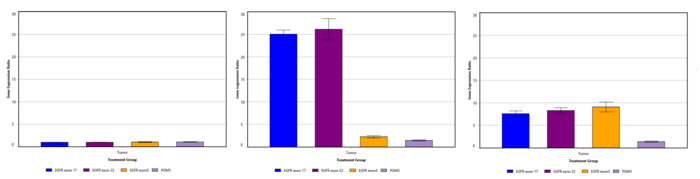
Abbildung: Quantitative Bestimmung des EGFR-Gens und dessen Variante EGFRvIII. Aus dem Verhältnis von Referenzgenen und Zielgen (Exone 3, 17, 22 von EGFR, sowie PGM5) aus Tumor und Blut wird bestimmt, ob eine EGFR-Amplifikation vorliegen. Bei einem Verhältnis von ~1 liegt keine Amplifikation vor (links), bei Werten deutlich über 2, ist das EGFR-Gen vervielfältigt (rechts). Ist Exon 3 (gelb) im Vergleich zu Exon 17 und 22 weniger stark vervielfältigt, liegt die EGFRvIII-Variante vor(Mitte).
Literatur:
- An et al. (2018) Epidermal growth factor receptor (EGFR) and EGFRvIII in glioblastoma (GBM): signaling pathways and targeted therapies. Oncogene. 37(12): 1561–1575.
- Brennan et al. (2013) The somatic genomic landscape of glioblastoma. Cell. 155:462–477.
- Eskilsson E et al. (2016) EGFRvIII mutations can emerge as late and heterogenous events in glioblastoma development and promote angiogenesis through Src activation. Neuro-oncology. 18:1644–1655.
- WHO Classification of Tumours, 5th Edition. Central Nervous System Tumours, Volume 6. WHO Classification of Tumours Editorial Board.ISBN-13 978-92-832-4508-7
- Bai and Wong (2005). Simultaneous detection and quantification of mitochondrial DNA deletion(s), depletion, and over-replication in patients with mitochondrial disease. J Mol Diagn. 7(5):613-22.
- Ma und Chung (2015) Quantitative Analysis of Copy Number Variants Based on Real-Time LightCycler PCR. Curr Protoc Hum Genet. 80: 7.21.1–7.21.8
- Cohen et al. (2015). DNA copy number analysis of Grade II–III and Grade IV gliomas reveals differences in molecular ontogeny including chromothripsis associated with IDH mutation status. acta neuropathol commun. doi.org/10.1186/s40478-015-0213-3