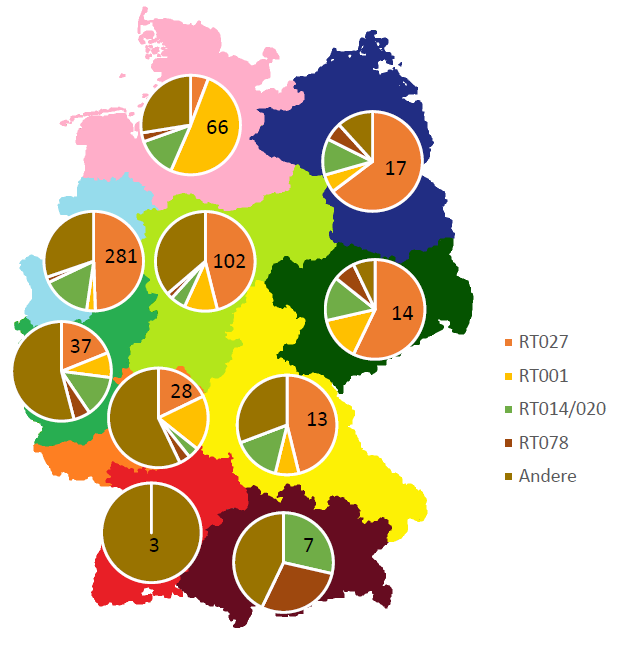
Prinzipiell kann jeder toxigene Stamm schwere Verlaufsformen verursachen. Insbesondere im Krankenhausumfeld sind die Ribotypen 001 (RT001) und RT027 weit verbreitet. RT027 ist ein Stamm, der ursprünglich aus Nordamerika stammt und erstmals im Jahr 2007 bei Ausbrüchen in Deutschland beschrieben wurde (1). Die Resistenz gegenüber Makroliden und Fluorochinolonen scheint hierbei einen wichtigen Selektionsfaktor darzustellen (2). Deutschlandweit gibt es starke regionale Prävalenzunterschiede, wobei diese 5.3% bis zu 33.5% betragen können (3,4,5). Darüber hinaus stets ein Eintrag weiterer Stämme aus dem Ausland erfolgen wie dem RT018 (6) oder dem RT176 (7). Insbesondere RT027 und RT001 lassen sich maßgeblich durch Antibiotic Stewardship (rationaler Antibiotikaeinsatz mit Verminderung der „4C“-Antibiotika: Clindamycin, Fluorochinolone, Cephalosporine und Aminopenicilline mit Betalaktamaseinhibitor) zurückdrängen (8). In der untenstehenden Abbildung finden Sie einen Überblick über die genotypischen Daten für die Jahre 2017 und 2018 basierend auf den zu uns gesandten Isolaten.
Isolate der Jahre 2017-2018 nach Einsendern und Postleitzahlenregionen (n=539). Es handelt sich dabei um Isolate vornehmlich aus Ausbrüchen und von schweren klinischen Verlaufsformen.
1. Kleinkauf N, Weiss B, Jansen A, Eckmanns T, Bornhofen B, Kuehnen E, et al. Confirmed cases and report of clusters of severe infections due to Clostridium difficile PCR ribotype 027 in Germany. Euro Surveill. 2007;12(11):E071115 2.
2. Freeman J, Bauer MP, Baines SD, Corver J, Fawley WN, Goorhuis B, et al. The changing epidemiology of Clostridium difficile infections. Clin Microbiol Rev. 2010;23(3):529-49.
3. von Müller L, Mock M, Halfmann A, Stahlmann J, Simon A, Herrmann M. Epidemiology of Clostridium difficile in Germany based on a single center long-term surveillance and German-wide genotyping of recent isolates provided to the advisory laboratory for diagnostic reasons. Int J Med Microbiol. 2015;305(7):807-13.
4. Arvand M, Hauri AM, Zaiss NH, Witte W, Bettge-Weller G. Clostridium difficile ribotypes 001, 017, and 027 are associated with lethal C. difficile infection in Hesse, Germany. Euro Surveill. 2009;14(45).
5. Arvand M, Bettge-Weller G. Clostridium difficile ribotype 027 is not evenly distributed in Hesse, Germany. Anaerobe. 2016;40:1-4.
6. Berger FK, Gfrörer S, Becker SL, Baldan R, Cirillo DM, Frentrup M, et al. Hospital outbreak due to Clostridium difficile ribotype 018 (RT018) in Southern Germany. Int J Med Microbiol. 2019.
7. Berger F, Bischoff M, Strauß C, von Müller L, Gärtner B. Auftreten von Clostridium difficile Ribotyp 176 (RT176) in Deutschland. Epid Bull. 2017(10:93 – 95).
8. Lawes T, Lopez-Lozano JM, Nebot CA, Macartney G, Subbarao-Sharma R, Wares KD, et al. Effect of a national 4C antibiotic stewardship intervention on the clinical and molecular epidemiology of Clostridium difficile infections in a region of Scotland: a non-linear time-series analysis. Lancet Infect Dis. 2017;17(2):194-206.